Centro de Investigación Científica
El Grupo de Inteligencia Artificial PUCP es reconocido desde mayo de 2017 como Centro de Investigación Científica por el Consejo Nacional de CIencia y Tecnología CONCYTEC y tiene áres fuertes en las disciplinas de Inteligencia Artificial, Aprendizaje de Máquina, Computación Gráfica, Minería de Datos, Visión Computacional y Procesamiento de Lenguaje Natural.
Fortalezas
Científica
Líneas de Investigación
-
Bioinformática
La bioinformática es una disciplina que permite aplicar herramientas de la informática al estudio y la gestión de datos de la biología. En su campo interactúan diversas áreas del conocimiento como las ciencias de la computación, la estadística y la química. Los principales esfuerzos de investigación en estos campos incluyen el alineamiento de secuencias, la predicción de genes, montaje del genoma, alineamiento estructural de proteínas, predicción de estructura de proteínas, predicción de la expresión génica, interacciones proteína-proteína, y modelado de la evolución.
-
Visión Computacional (2D y 3D)
Comprende el análisis y la interpretación de la información visual. La comprensión de la imagen se considera como un proceso que parte en una imagen o secuencia de imágenes (por ejemplo, proyecciones 2D de una escena estática o dinámica) y termina en una descripción interna de la escena. Los problemas de la interpretación de imágenes son el núcleo de los esfuerzos actuales para permitir hacer una máquina que tenga interacciones "inteligentes" con su entorno.
-
Procesamiento de Lenguaje Natural
Tiene como objetivo conseguir que las computadoras procesen el lenguaje humano en sus diferentes niveles, como el morfológico, sintáctico o semántico. A partir de ello, se pueden desarrollar aplicaciones de diversa complejidad, desde un corrector ortográfico hasta un traductor automático.
-
Ingeniería del Conocimiento
La ingeniería del conocimiento forma parte de la Inteligencia Artificial y su objetivo es diseñar y desarrollar de Sistemas Expertos intentando representar el conocimiento y razonamiento humanos en un determinado dominio.
Proyectos Actuales
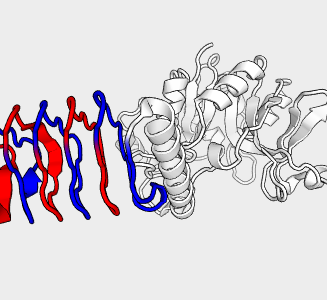
Análisis y predicción de proteinas repetidas
Las proteínas repetidas son una clase extendida de proteínas no globulares que llevan a cabo funciones heterogéneas involucradas en varias enfermedades.
Traducción automática híbrida (MT) para las lenguas indígenas peruanas
Traducción automática de texto entre lenguas nativas de la amazonía Peruana y el español.
Análisis de la simetría y restauración de Objectos 3D
Herramientas computacionales que facilitan la conservación y restauración de material del patrimonio cultural. Simetría como una especificación de estructura.

Detección de causalidad en datos genómicos
La detección de las interacciones génicas causales a partir de perfiles de expresión génica temporal es un tema clave en genómica, ya que podría reducir la necesidad de ensayos intervencionistas.

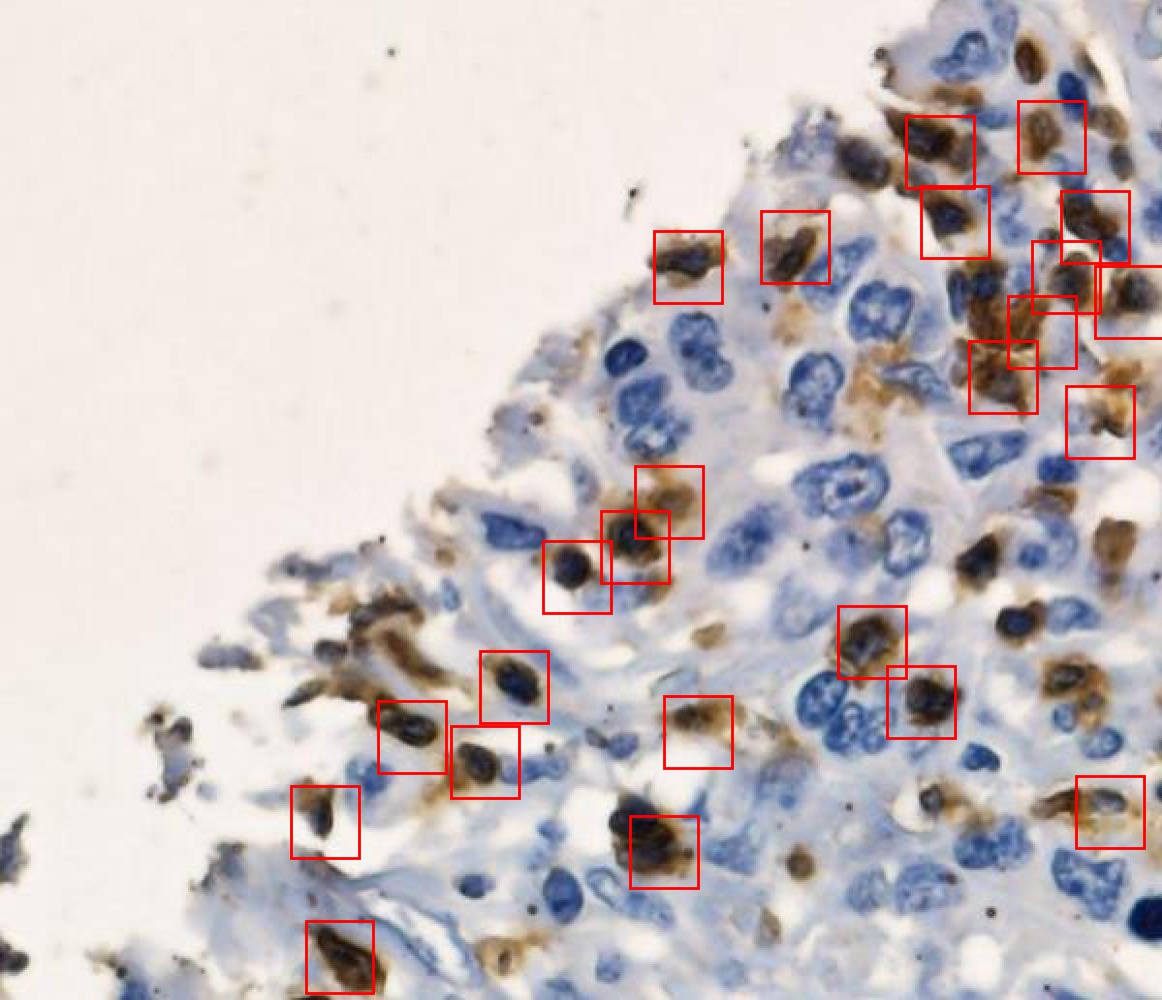
Detección Automática de Células
Enfoque usando Deep Learning para detectar y contar linfocitos tumor-infiltrados en imágenes inmunohistoquímicas de Cáncer Gástrico.
Plataforma para anotación semántica y visualización de documentos web
Los documentos en la web requieren herramientas especiales para realizar una búsqueda eficiente, pero están distribuidos en diferentes dominios.
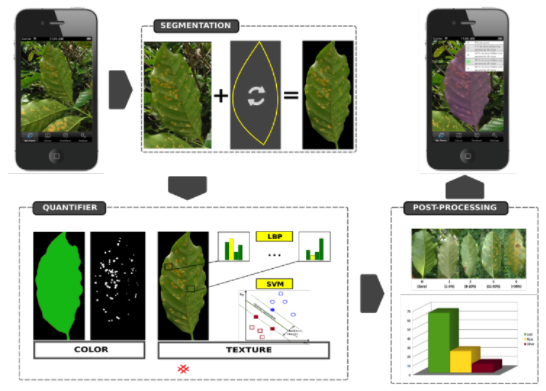
Diagnóstico automático de la enfermedad de la roya del café
Aplicación de técnicas de procesamiento de imágenes para el diagnóstico automático de enfermedades en cultivos de café.
Evaluación automática de fertilidad masculina
Aplicación de procesamiento de imágenes para el análisi automatico de características de las células espermáticas, como la concentración, la motilidad y la morfología.

Publicaciones
-
ChAnot: An intelligent annotation tool for indigenous and highly agglutinative languages in Peru
Mercado, R., Pereira, J., Sobrevilla-Cabezudo, M. & Oncevay-Marcos, A. (2018). In Proceedings of the Eleventh International Conference on Language Resources and Evaluation (LREC 2018). (In-press)
-
WordNet-Shp: Towards the building of a lexical database for a Peruvian minority language
Maguiño, D., Oncevay-Marcos, A. & Sobrevilla-Cabezudo, M. (2018). In Proceedings of the Eleventh International Conference on Language Resources and Evaluation (LREC 2018). (In-press)
-
Corpus building and evaluation of aspect-based opinion summaries from tweets in Spanish
Peñaloza, D., López, R., Tenorio, J., Gómez, H., D., Oncevay-Marcos, A. & Sobrevilla-Cabezudo, M. (2018). In Proceedings of the Eleventh International Conference on Language Resources and Evaluation (LREC 2018). (In-press)
-
Language identification with scarce data: A case study from Peru
Espichán-Linares, A. & Oncevay-Marcos, A. (2018). In Information Management and Big Data: Fourth Annual International Symposium, SIMBig 2017, Lima, Peru, September 4-6, 2017, Revised Selected Papers. Springer International Publishing. (In-press)
-
Classification of β-hairpin repeat proteins.
Roche, D.; Do Viet, P.; Bakulina, A.; HIRSH, L.; Tosatto, S. y Kajava, A. (2017). Elsevier Editorial System(tm) for Journal of Structural Biology.
-
Spell-checking based on syllabification and character-level graphs for a Peruvian agglutinative language
Alva, C. & Oncevay-Marcos, A. (2017). In Proceedings of the EMNLP 2017 Workshop on Subword and Character Level Models in NLP, SCLeM 2017. ACL Anthology.
-
Corpus creation and initial SMT experiments between Spanish and Shipibo-konibo
Galarreta, A. P., Melgar, A., & Oncevay-Marcos, A. (2017). In Proceedings of the International Conference on Recent Advances in Natural Language Processing, RANLP 2017.
-
Exploratory analysis for ontology learning of events on social media streaming in Spanish
Valeriano, E. & Oncevay-Marcos, A. (2017). In Proceedings of the IWCS 2017 Workshop on Language, Ontology, Terminology and Knowledge Structures, LOTKS 2017. ACL Anthology.
-
Ship-LemmaTagger: building an NLP toolkit for a Peruvian native language
Pereira, J., Mercado, R., Melgar, A., Sobrevilla-Cabezudo, M., & Oncevay-Marcos, A. (2017). In Text, Speech, and Dialogue: 20th International Conference, TSD 2017, Prague, Czech Republic, August 27-31, 2017, Proceedings (pp. 473-481). Springer International Publishing.
-
SenseDependency-Rank: A word sense disambiguation method based on random walks and dependency trees
Sobrevilla-Cabezudo, M., Oncevay-Marcos, A., & Melgar, A. (2017). In Computational Linguistics and Intelligent Text Processing: 18th International Conference, CICLing 2017, Budapest, Hungary, April 17-23, 2017, Proceedings. Springer International Publishing. (In-press)
-
Scalable 3d shape retrieval using local features and the signature quadratic form distance
Sipiran, I., Lokoc̆, J., Bustos, B., & Skopal, T. (2017). The Visual Computer, 33(12), 1571-1585.
-
From reassembly to object completion: A complete systems pipeline
Papaioannou, G., Schreck, T., Andreadis, A., Mavridis, P., Gregor, R., Sipiran, I., & Vardis, K. (2017). Journal on Computing and Cultural Heritage (JOCCH), 10(2), 8.
-
Automatic Lymphocyte Detection on Gastric Cancer IHC Images Using Deep Learning
E. Garcia, R. Hermoza, C. B. Castanon, L. Cano, M. Castillo and C. Castaneda, 2017 IEEE 30th International Symposium on Computer-Based Medical Systems (CBMS), Thessaloniki, 2017, pp. 200-204.
-
Analysis of Partial Axial Symmetry on 3D Surfaces and Its Application in the Restoration of Cultural Heritage Objects.
Sipiran, I. (2017). In The IEEE International Conference on Computer Vision (ICCV) (Vol. 2).
-
3D Reconstruction of Incomplete Archaeological Objects Using a Generative Adversary Network
Hermoza, R., & Sipiran, I. (2017). arXiv preprint arXiv:1711.06363.
-
Identification of repetitive units in protein structures with ReUPred
HIRSH, L.; Piovesan, D.; Paladin, L. y Tosatto, S. (2016). Amino Acids, 48 (6), pp. 1391-1400.
-
RepeatsDB 2.0: improved annotation, classification, search and visualization of repeat protein structures
Paladin, L.; HIRSH, L.; Piovesan, D.; Andrade, M.; Kajava, A. y Tosatto, S. (2016). Nucleic Acids Research, d1 (45), pp. 308-312.
-
Guiding the Exploration of Scatter Plot Data Using Motif-based Interest Measures
Shao, L., Schleicher, T., Behrisch, M., Schreck, T., Sipiran, I., & Keim, D. A. (2016). Journal of Visual Languages & Computing, 36, 1-12.
-
Coh-Metrix-Esp: A Complexity Analysis Tool for Documents Written in Spanish
Quispersaravia, A., Perez, W., Sobrevilla, M., & Alva-Manchengo, F. (2016). In Proceedings of the Tenth International Conference on Language Resources and Evaluation (LREC 2016).
